Command Palette
Search for a command to run...
Ping Xu Zhiyuan Ning Pengjiang Li Wenhao Liu Pengyang Wang Jiaxu Cui Yuanchun Zhou Pengfei Wang
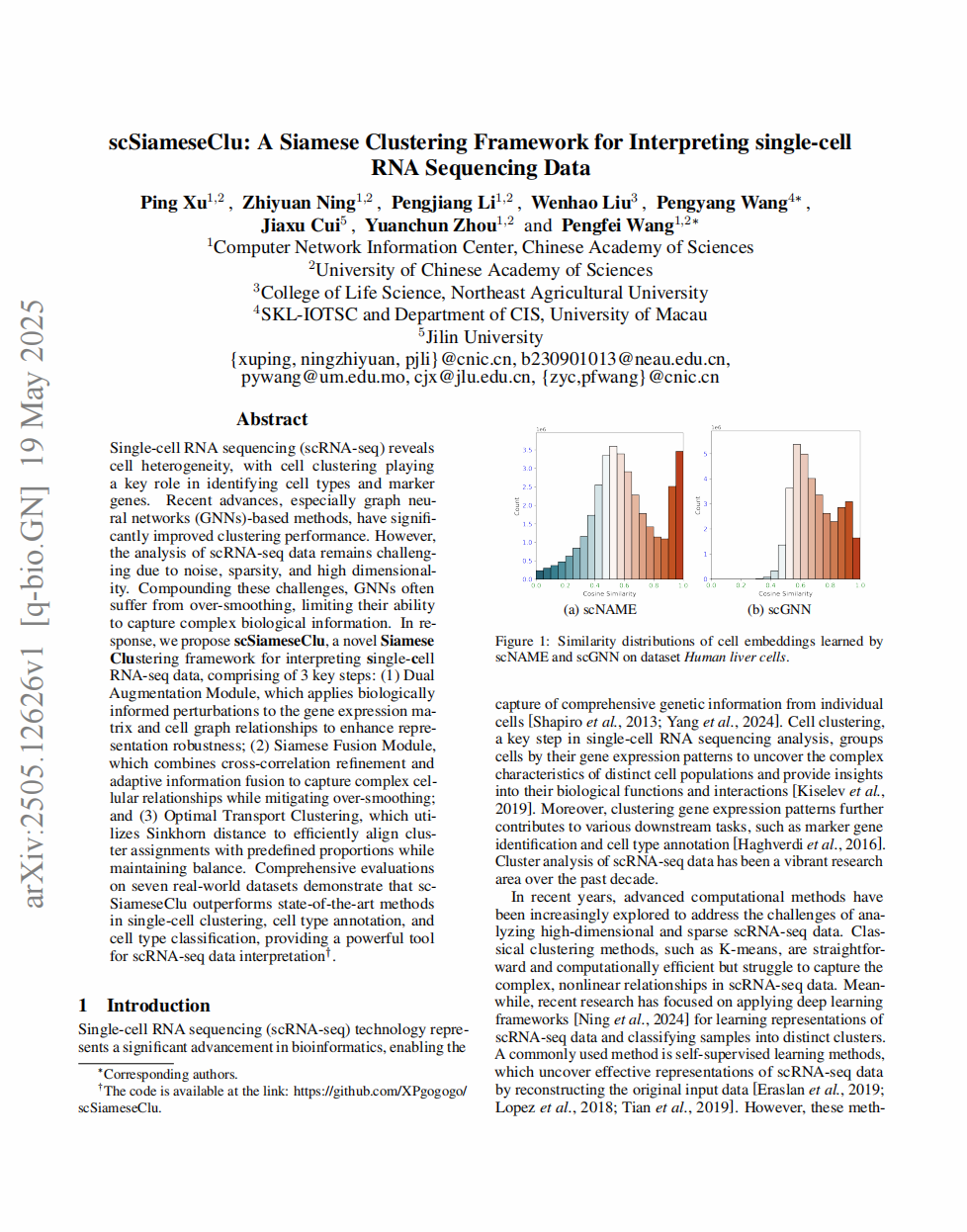
摘要
单细胞RNA测序(scRNA-seq)能够揭示细胞异质性,其中细胞聚类在识别细胞类型和标记基因方面发挥着关键作用。近年来,特别是基于图神经网络(GNNs)的方法在提升聚类性能方面取得了显著进展。然而,由于数据中普遍存在噪声、稀疏性以及高维度等问题,scRNA-seq数据的分析仍面临巨大挑战。此外,GNN模型常遭遇过平滑(over-smoothing)问题,限制了其捕捉复杂生物学信息的能力。针对上述问题,我们提出了一种名为scSiameseClu的新颖Siamese聚类框架,用于解释单细胞RNA测序数据,该框架包含三个核心步骤:(1)双增强模块(Dual Augmentation Module),通过施加具有生物学意义的扰动于基因表达矩阵及细胞图结构关系,提升表征的鲁棒性;(2)Siamese融合模块(Siamese Fusion Module),结合交叉相关性精炼与自适应信息融合机制,在捕捉复杂细胞间关系的同时有效缓解过平滑问题;(3)最优传输聚类(Optimal Transport Clustering),利用Sinkhorn距离高效地将聚类结果与预设的细胞类型比例对齐,同时保持类别分布的平衡性。在七个真实世界数据集上的全面评估表明,所提出的~\methodname~在单细胞聚类、细胞类型注释及细胞类型分类任务中均优于现有最先进方法,为scRNA-seq数据的深入解析提供了一个强大而有效的工具。